LACA是用于长扩增子一致性分析(例如 16S rRNA 基因扩增子分析)的可重复且可扩展的工作流程。它使用用snakemake管理工作流程以及conda来管理环境。
LACA 的安装
完整的安装指南LACA可在此处获取。
您可以根据您的喜好选择使用docker或从GitHub存储库安装LACA
Docker 镜像
最简单的使用方法是从Docker Hub拉取LACA镜像以获得跨平台支持
1 | docker pull yanhui09/laca |
LACA是通过docker为linux/amd64平台而构建的,
MacOS用户需要使用 docker 容器来运行LACA。
从 GitHub 存储库安装
1.克隆 Github 仓库并创建隔离conda环境
1 | git clone https://github.com/yanhui09/laca.git |
2.安装LACA
为了避免不一致,建议在上面建立的conda环境中安装LACA
1 | conda activate laca |
使用 LACA 运行演示数据
在这里找到完整的使用指南。
快速启动示例
1 | laca init -b /path/to/basecalled_fastqs -d /path/to/database # init config file and check |
熟悉LACA使用
LACA很容易使用。您可以使用laca init和laca run分两步开始新的分析。
如果LACA安装在conda环境中,请记住激活conda环境。
1 | conda activate laca |
要使用 docker 镜像,您需要将数据目录(例如pwd)挂载到容器中/home 目录。
1 | docker run -it -v `pwd`:/home --privileged yanhui09/laca |
1.初始化配置文件laca init
laca init会在工作目录中生成一个配置文件,其中包含运行LACA所需的所有参数。
1 | laca init -h |
2.开始laca run分析
laca run将相应地触发完整的工作流程或定义资源下的特定模块。使用laca run -h获得试运行概述。
1 | laca run -h |
使用演示数据集运行LACA
0.确保您已从此处下载所需的演示数据集。然后cd进入目录。
例如,输入绝对路径(“长路径”)/home/me/MAC2023-extra。
1 | cd /home/me/MAC2023-extra |
如果您尚未下载数据用Git下载,
1 | git clone https://github.com/yanhui09/MAC2023-extra.git |
1.检查您所在的位置并尝试laca init检查生成的config.yaml文件。
1 | pwd |
2.LACA伪运行和真实运行
1 | laca run all -w ./laca_output -n |
LACA能够生成otu table,taxonomy table以及phylogenetic tree如果您使用laca run all运行完整的工作流程。但第一次使用需要时间准备数据库和安装。
作为一个例子,这里我们只运行模块kmerCon来根据 kmer 频率提取一致序列。
看看这些共有序列,取第一个序列对rRNA/ITS数据库进行BLAST搜索。
1 | head -n2 ./laca_output/kmerCon/kmerCon.fna |
预期输出:
1 | >pooled_0b000_0cand1 |
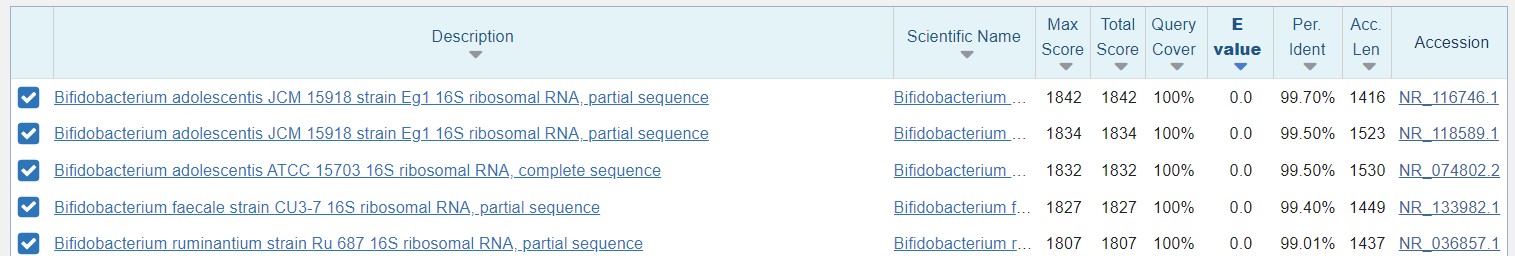
结果BLAST表明,该序列是一个16S rRNA基因片段,Bifidobacterium一性性超过 99%。
读取一个 ONT 并进行相同的
BLAST搜索。您希望看到什么?
本文为学习记录,课程作者为:YanHui